Le manque d'interprétabilité de l'apprentissage profond entrave son utilisation en médecine qui exige la transparence des décisions. Par exemple, un modèle de classification (sain versus pathologique) devrait s'appuyer sur des marqueurs radiologiques et non sur des biais potentiels de la base de données d'apprentissage.
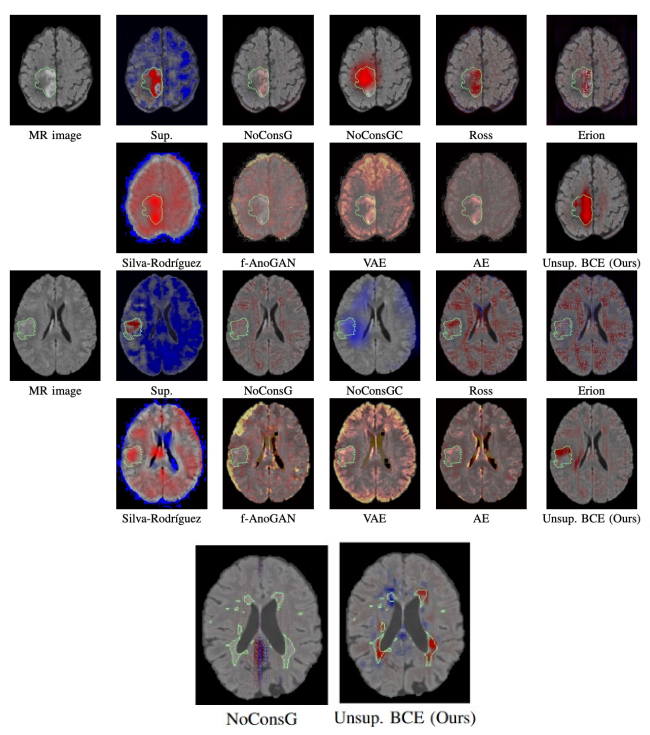
Dans cet article, nous proposons une nouvelle méthode faiblement supervisée pour une classification (sain/pathologique) interprétable et pour la détection d'anomalies. Une nouvelle fonction de perte est ajoutée à un modèle de classification standard pour contraindre chaque voxel (des images saines) à orienter la décision du réseau vers la classe saine selon des attributions fondées sur le gradient. Cette contrainte révèle des structures pathologiques pour les images de patients, permettant leur segmentation non supervisée. Nous proposons également une combinaison d'attributions au cours de l'entraînement contraint, ce qui rend le modèle plus robuste.
Notre approche a été évaluée sur deux pathologies cérébrales, en cancérologie et en sclérose en plaques. Cette nouvelle contrainte fournit une classification plus pertinente, avec une décision plus orientée vers la pathologie. Trois jeux de données publics FLAIR IRM ont été utilisés pour les images « saines » : MPI, kirby21 et IBC. Les images de tumeurs cérébrales proviennent de BraTS 2019 et 2020. MICCAI MSSEG 2016 et le jeu de données OFSEP/EDMUS1 de l' « Observatoire français de la sclérose en plaques » ont été utilisés pour les images de sclérose en plaques. Les images ont été acquises dans différents centres avec des scanners de divers constructeurs de 1,5T et 3T. En comparaison avec de nombreuses méthodes, notre méthode s’avère la plus cohérente avec les a priori clinique. Pour la détection d'anomalies, la méthode proposée surpasse l'état de l'art, en particulier sur la tâche difficile de segmentation des lésions de sclérose en plaques.
Notre réseau a été implémenté avec Pytorch. Le code source est disponible sur GitHub en cliquant ici.