2019/2020 : Offre de stage PFE/Master 2:
Segmentation d’images médicales 3D par transfert de points clés
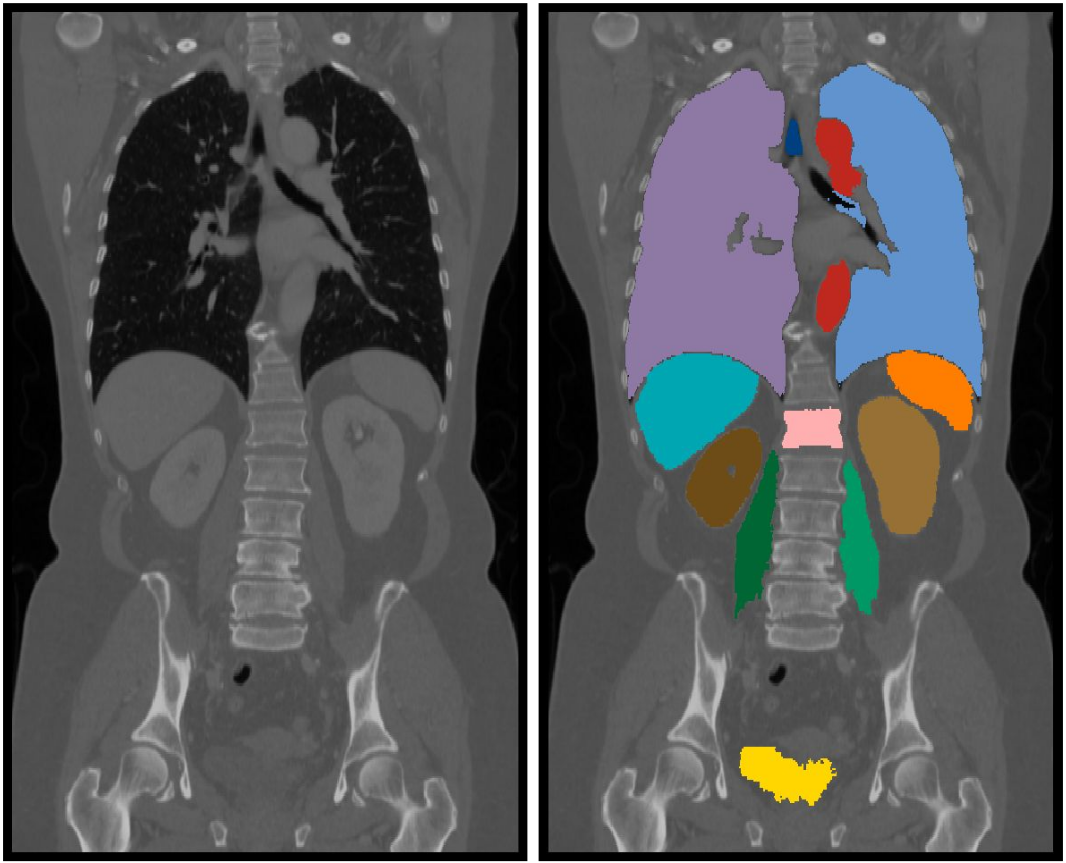
Figure 1 : Segmentation d’une image CT médicale. Gauche : image originale, droite : organes segmentés.
Contexte
Le projet ANR TOPACS ( https://www.creatis.insa-lyon.fr/~valette/public/project/topacs/ ) vise à étudier la pertinence des points clés 3D pour l’analyse de grande masses d’images médicales 3D. Un point clé décrit une région singulière d’une l’image à l’aide d’un descripteur (vecteur caractéristique). Un ensemble de points clés extraits d’une image constitue un résumé efficace de l’image. Une telle représentation offre plusieurs avantages : données compactes, réduction du coût calculatoire pour diverses opérations, comme le recalage de grands groupes d’images [1] et la segmentation [2] (voir Figure 1) .
Objectif
L’objectif du stage est l’étude de faisabilité de l’approche de segmentation par transfert de points clés proposée par Wachinger et al. [2]. Nous disposons à l’heure actuelle de plusieurs bases de données images annotées qui serviront de référence pour la segmentation, de plusieurs outils d’extraction de points clés (SURF3D, SIFT3D), et d’outils de mise en correspondance.Le travail du stagiaire sera donc concentré sur le transfert de points clés. Dans un premier temps, le transfert sera réalisé entre des images de même modalité, et en fonction du déroulement du stage, un transfert entre modalités différentes pourra être expérimenté, en utilisant par exemple des descripteurs invariants à la modalité [3], ou une méthode d’apprentissage par transfert [4].
Déroulement du stage
Le stage se déroulera au laboratoire CREATIS, et sera encadré par
- Razmig Kéchichian (MCF INSA/Creatis) razmig.kechichian@creatis.insa-lyon.fr
- Michel Desvignes (PR Grenoble INP/GIPSA-lab) michel.desvignes@grenoble-inp.fr
- Sébastien Valette (CR CNRS/Creatis) sebastien.valette@creatis.insa-lyon.fr
Profil recherché
Nous recherchons une personne ayant des compétences en mathématiques appliquées, traitement d’images et informatique.
En fonction du déroulement du stage, une poursuite en thèse pourra être envisagée pour le ou la candidate.
Candidature
Nous encourageons les candidats intéressés à nous envoyer un CV accompagné d'une lettre de motivation et d’un relevé de notes récent.
Gratification
Le stagiaire sera rémunéré au taux fixé par la loi: ~ 540 € / mois.
Références
[1] R. Agier, S. Valette, R. Kéchichian, L. Fanton, R. Prost, Hubless keypoint-based 3D deformable groupwise registration, Medical Image Analysis, à paraître.https://arxiv.org/abs/1809.03951
[2] C. Wachinger, M. Toews, G. Langs, W. Wells, P. Golland, Keypoint transfer for fast whole-body segmentation, IEEE TMI, 2018 . https://arxiv.org/abs/1806.08723
[3] M. P. Heinrich, M. Jenkinson, M. Bhushanab, T. Matin, F. V. Gleesond, M. Brady, J. A.Schnabela, MIND: Modality independent neighbourhood descriptor for multi-modal deformable registration, Medical Image Analysis , Volume 16, no 7, Octobre 2012.
[4] J.-Y. Zhu, T. Park, P. Isola, A.A Efros, Unpaired image–to–image translation using cycle–consistent adversarial networks, IEEE international conference on computer vision, 2017, pp. 2223–2232.
