Les nombreux phénomènes physiologiques actifs et passifs qui ont lieu au cours d'un cycle cardiaque sont intimement liés à la structure complexe du coeur. Bien que l'architecture du coeur soit sujet à controverse, notamment à cause du manque de données in-vivo et 3D, les nouvelles techniques d'imagerie ainsi que les modèles permettent une meilleure compréhension de la morphologie, la physiologie et les aspects cliniques des maladies cardiovasculaires, et permettent notamment d'aider à la détermination d'approches thérapeutiques optimales.
Jusqu'à maintenant, à cause du manque de méthodes non invasives, l'étude in-vivo de l'architecture fibreuse du ventricule gauche en réponse aux déformations ayant lieu durant la systole et la diastole ne pouvait se faire de manière intensive. Nous avons axé nos recherches afin de répondre à ces problématiques.
Axes de recherche
Nos projets sont:
IRM cardiaque du tenseur de diffusion in-vivo
Pour réaliser une technique DT-MRI in-vivo robuste en respiration libre, applicable en protocole clinique et expérimental (collaboration NIH, H.Wen; Thèse S. Rappachi). Nous avons obtenu des résultats préliminaires in-vivo avec des acquisitions en respiration libre, qui amènent de nouvelles perspectives en vue du DT-MRI in-vivo clinique, même pour des patients en mauvaise condition.
Impact de la régularisation sur des données in-vivo. De gauche à droite : cartes paramétriques MD, FA, champ PDD. De haut en bas : paramètres calculés à partir des images temporal Maximum Intensity Projection(tMIP)-DW brutes, puis régularisées
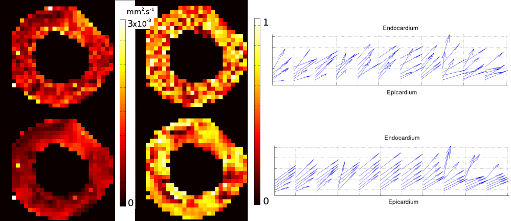
Impact de la régularisation sur des données in-vivo.
Prise en compte du mouvement via tMIP-DWI, et premiers résultats sur l'être humain. Le but est ici de développer une approche DWI qui tienne compte du mouvement, ainsi que de démontrer sa faisabilité en routine clinique
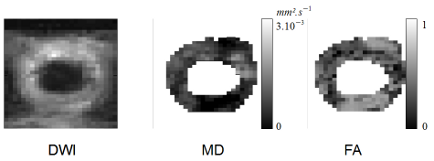
Résultats sur volontaire sain : DWI, diffusion moyenne, carte d'anisotropie fractionnelle.
Modélisation patient-spécifique
Pour créer un modèle patient spécifique à partir d'une imagerie multi-modalité, afin d'intégrer dans un modèle unique la forme (anatomie), la structure (propriétés des tissus, architecture fibreuse) et le mouvement (dynamique) du coeur.
Segmentation 3D statique en utilisant le modèle éléments finis DET3 (Deformable Elastic Template 3D). Ce modèle est un modèle volumétrique élastique soumis à des forces extraites de l'image
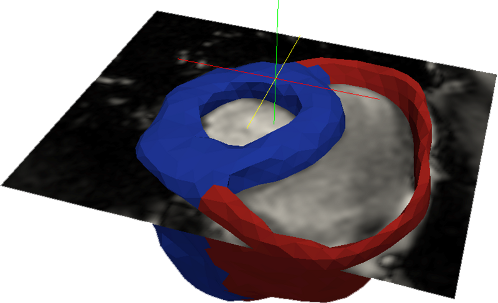
Segmentation 3D statique du coeur.
Segmentation 2D dynamique en utilisant le modèle Dynamic DET. Il s'agit d'une extension spatio-temporelle du modèle DET, contrôlée par une équation de la Dynamique simplifiée

Segmentation 2D dynamique du ventricule gauche.
Applications cliniques
Pour mieux comprendre, en utilisant des modèles de coeur patient-spécifiques, les changements structurels et fonctionnels qui ont lieu dans plusieurs cardiomyopathies au cours des processus de remodélisation.

