- Participants
Nicolas Duchateau, Maxime Di Folco, Benoit Freiche, Patrick Clarysse
- Collaborations nationales et internationales
Universitat Pompeu Fabra, Barcelone, ES (Sergio Sanchez, Gemma Piella, Bart Bijnens)
CHU Nice, FR (Pamela Moceri)
- Question
Comment intégrer un grand nombre de données, souvent hétérogènes et complexes ? Quel impact pour la compréhension des pathologies ?
- Objectif
Approfondir des méthodes d’apprentissage sur des données hétérogènes en haute dimension, et les utiliser pour (i) mieux représenter la distribution d’une population, et (ii) évaluer l’importance relative des descripteurs utilisés, et mettre en évidence de nouvelles interactions entre ces descripteurs.
- Méthode
La méthodologie se base sur des algorithmes d'apprentissage de variété non-supervisés qui permettent également de réduire la dimension des données. La variabilité des données s’étudie dans l’espace de moindre dimension ou après reconstruction dans l’espace d’entrée, selon les besoins de l’application.
Nous avons dans un premier temps contribué au développement d'une version non-supervisée de l’algorithme d’apprentissage à noyaux multiples (Multiple Kernel Learning [Lin:2011]). Une matrice d’affinité est construite pour chaque descripteur à l’aide d’un noyau adéquat, et l’algorithme optimise de façon conjointe le poids relatif accordé à ces descripteurs et une matrice de changement de base vers un espace de moindre dimension plus approprié. Nous étudions également une stratégie permettant de mieux prendre en compte le lien entre les descripteurs utilisés en entrée, et notamment leur corrélation et redondance potentielles [DiFolco:2019].
- Résultats et illustrations
Nous avons testé la méthodologie à noyaux multiples sur différents descripteurs des vitesses myocardiques issus d’acquisitions d'échocardiographie tissue Doppler pendant un protocole d’effort afin de mieux caractériser le syndrome d’insuffisance cardiaque à fraction d’éjection préservée [Sanchez:2017, Sanchez:2018], ainsi que sur un panel de descripteurs cliniques et issus d'échocardiographie 2D afin d'identifier des différences de réponse à la thérapie de resynchronisation cardiaque [Cikes:2019].
Le lien entre descripteurs est actuellement exploré dans le cadre de la forme et de la déformation cardiaques, pour l'étude de la surcharge en pression et en volume du ventricule droit avec des données d'échocardiographie 3D.
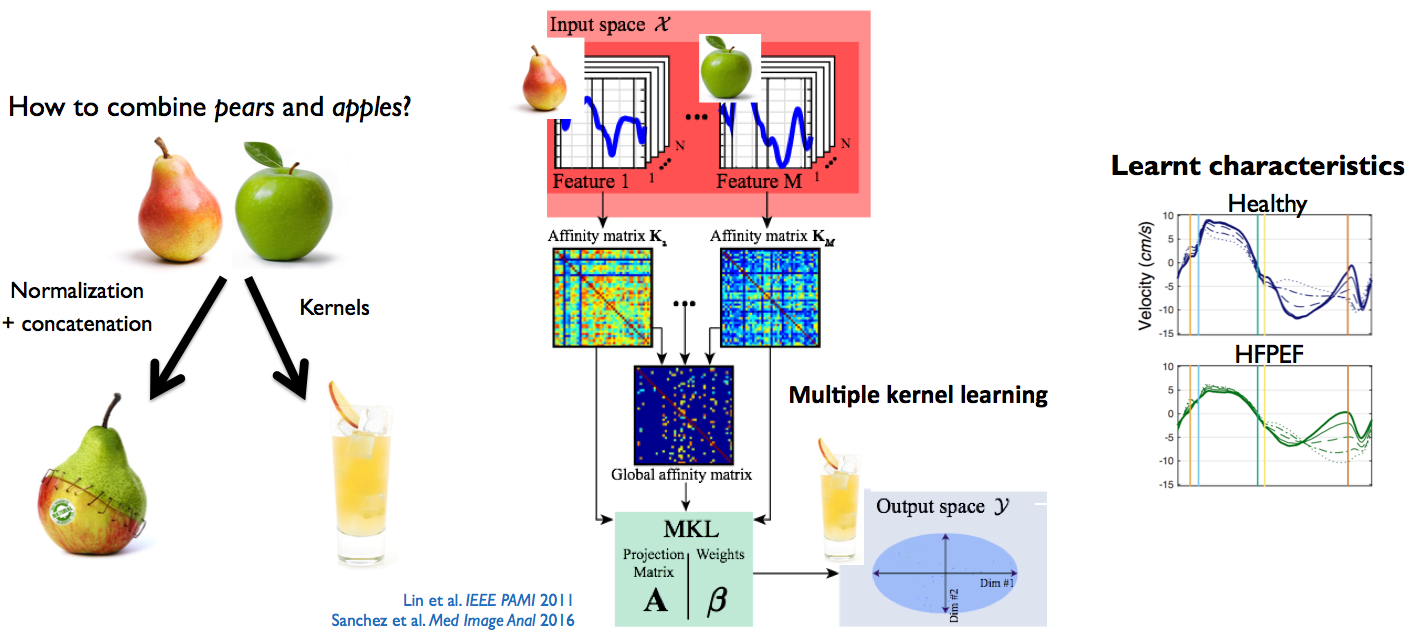
Figure 1: méthodologie employée pour mieux représenter les données issues de plusieurs descripteurs hétérogènes. Illustration sur des vitesses myocardiques de sujets normaux et avec insuffisance cardiaque à fraction d’éjection préservée, avant et pendant un protocole d’effort. Adapté de [Sanchez:2017]
- Soutiens financiers
PALSE_IMPULSION_2017 (Programme d’Avenir Lyon Saint-Etienne).
VP2HF (FP7-ICT-2013-611823).
ANR JCJC MIC-MAC (ANR-19-CE45-0005).
- Références bibliographiques
[Lin:2011] Lin YY, Liu TL, Fuh CS. Multiple kernel learning for dimensionality reduction. IEEE Trans Pattern Anal Mach Intell. 2011;33:1147-60.
[Sanchez:2017] Sanchez-Martinez S, Duchateau N, Erdei T, Fraser A, Bijnens B, Piella G. Characterization of myocardial motion patterns by unsupervised multiple kernel learning. Med Image Anal. 2017;35:70-82.
[Sanchez:2018] Sanchez-Martinez S, Duchateau N, Erdei T, Kunszt G, Aakhus S, Degiovanni A, Marino P, Carluccio E, Piella G, Fraser AG, Bijnens BH. Machine learning analysis of left ventricular function to characterize heart failure with preserved ejection fraction. Circ Cardiovasc Imaging. 2018;11:e007138.
[Cikes:2019] Cikes M, Sanchez-Martinez S, Claggett B, Duchateau N, Piella G, Butakoff C, Pouleur AC, Knappe D, Biering-Sørensen T, Kutyifa V, Moss A, Stein K, Solomon SD, Bijnens B. Machine-learning based phenogrouping in heart failure to identify responders to resynchronization therapy. Eur J Heart Fail. 2019;21:74-85.
[DiFolco:2019] Di Folco M, Clarysse P, Moceri P, Duchateau N Learning interactions between cardiac shape and deformation: application to pulmonary hypertension. Proc. STACOM-MICCAI, LNCS. 2019. In press.

