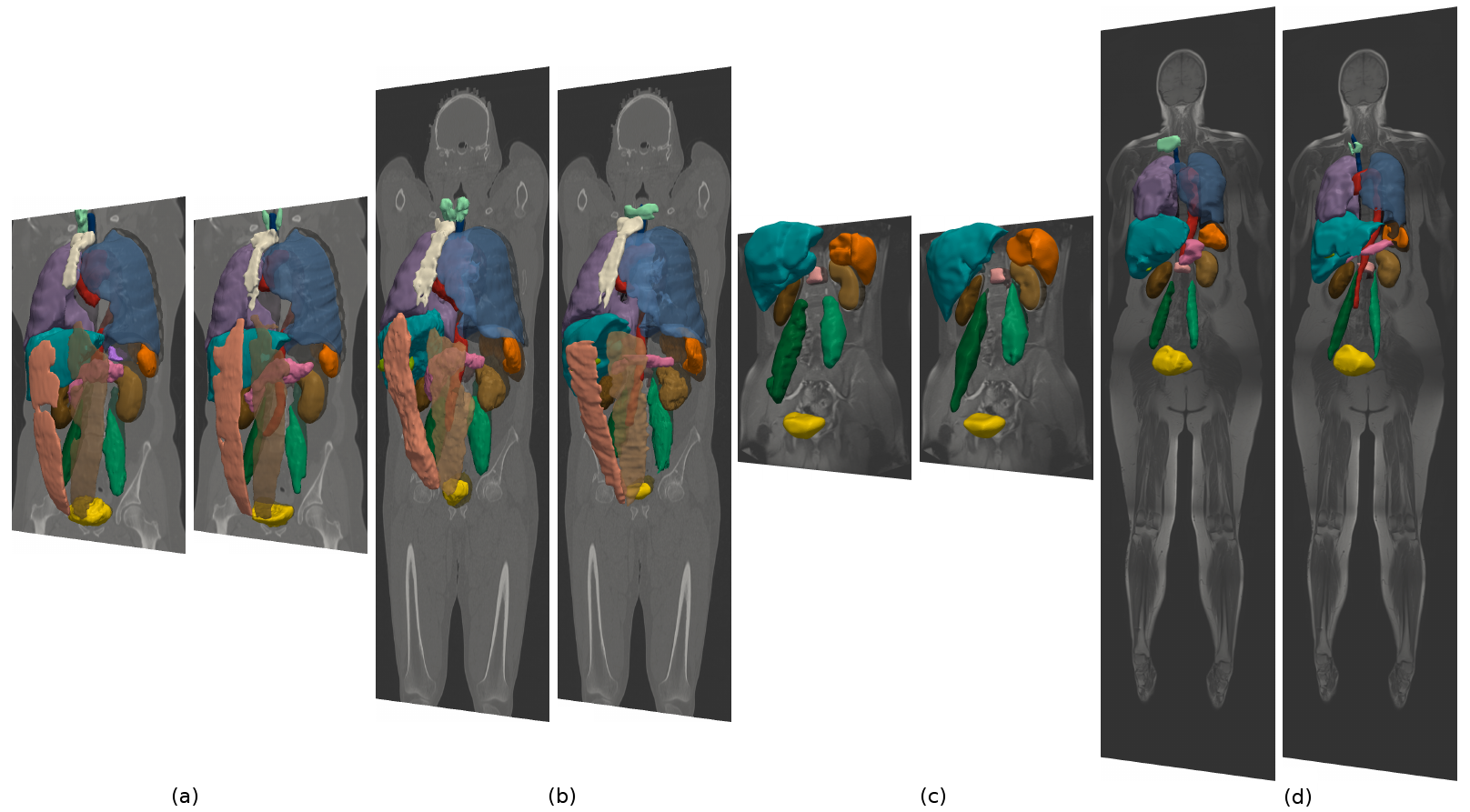
Illustration 1: Vues 3D des maillages surfaciques extraits des volumes de structures segmentées par notre méthode de segmentation multi-objet semi-automatique. Dans chaque paire, l’image de gauche correspond à la segmentation, l’image de droite à la référence. (a) TDM thoracique-abdominale contrastée, (b) TDM non contrastée,
(c) IRM abdominale contrastée et (d) IRM non contrastée.
Introduction
La pratique clinique et la recherche médicale génèrent chaque jour des milliers d’images médicales 3D rendant difficile les tâches d’accès, d’analyse et de visualisation, particulièrement dans les environnements distribués, mobiles et web. Il s’agit du phénomène « Big Data ». La segmentation, c'est-à-dire, l’extraction de structures anatomiques dans les images est souvent une condition préalable à leur interprétation et visualisation efficaces. L’apprentissage automatique, un domaine en plein essor aujourd’hui, fournit des méthodes d’extraction de caractéristiques et de définition de métriques permettant de catégoriser les structures apparaissant dans les images. Le vaste champ applicatif de ces méthodes couvre de nombreuses applications telles que l’aide au diagnostic, la radiothérapie, le planning pré-opératoire, la création de modèles anatomiques patient-spécifiques, la recherche d’images par le contenu, la création de prothèses sur-mesure (maintenant à l’aide de l’impression 3D), l’éducation, etc.
Contexte général et objectifs
Le but de ce stage est l’exploration des méthodes d’apprentissage automatique dans le contexte de la segmentation automatique d’images médicales 3D multi-modalité de grande taille et diversité. Il visera à une extraction plus robuste des connaissances et leur exploitation afin de rendre une méthode de segmentation générique développée au laboratoire Creatis, se limitant actuellement à des mesures photométriques élémentaires, plus performante en s’auto-adaptant, par ailleurs, à la modalité et à l’anatomie imagée. Réciproquement, les travaux du stage permettront de proposer de nouvelles approches d’extraction de caractéristiques et des stratégies d’apprentissage de métriques adaptées à des données peu traitées jusqu’ici par ces méthodes.
Contexte technique
La piste privilégiée sera une méthode de segmentation multi-objet automatique générique pour les images médicales 3D développée au laboratoire Creatis [KECH-14]. La segmentation est posée comme un problème d’étiquetage bayésien, faisant intervenir des relations spatiales entre les organes [KECH-13] et des vraisemblances (probabilités) d'intensité et de position pour chaque organe. La segmentation optimale est obtenue à l’aide d'un algorithme d’optimisation par coupure de graphes [BOYK-01]. L'illustration 1 affiche quelques résultats produits par cette méthode sur des images appartenant à 4 modalités différentes issues de la base de données Visceral [JIME-16].
Démarche et originalité
Les vraisemblances d’intensité, se limitant actuellement dans notre approche aux simples histogrammes d’intensité, peuvent être mieux estimées à partir des descripteurs d’intensité et de texture à l’aide des méthodes d’apprentissage automatique performantes comme les forêts aléatoires ou les réseaux de neurones [BISH-06]. Cela nécessite une extraction et sélection automatiques des descripteurs adaptées à la modalité de l'image cible et à l’organe. Des méthodes d’apprentissage automatique ayant des performances excellentes pour des images naturelles pourraient être adaptées et appliquées aux images médicales. Une première approche serait d’apprendre automatiquement des modèles de textures ou de formes locales à partir des voxels en utilisant des descripteurs classiques comme des histogrammes de gradients orientés (HOG) ou des « Local Binary Pattern » (LBP).Une deuxième approche consisterait à apprendre les descripteurs avec un réseau de neurones à convolution, soit de manière supervisée ou de manière non-supervisée/supervisée mixte [ZHEN-15]. Les travaux de recherche seront poursuivis sur la base de données multi-modalité Visceral [JIME-16] de taille relativement modeste ; elle offre 80 images accompagnées des 20 annotations d’organe. Les possibilités d’application non supervisée sur une base de données plus grande (plus de 150 patients) fournie par les praticiens hospitaliers du laboratoire Creatis pourront être étudiées. Les approches de l’état de l’art en segmentation automatique multi-organe se focalisent majoritai-rement sur les images tomodensitométriques [JIME-16, TONG-15], tandis que celles faisant intervenir les méthodes d’apprentissage automatique [KOHL-11, SEIF-09] exploitent des descripteurs prédéfinis et s’appuient dans l’estimation des métriques sur des bases de données d’apprentissage de taille importante. L'originalité de l'approche proposée sera donc la capacité de s’adapter à plusieurs modali-tés et à plusieurs organes par modalité, et ce avec peu de données annotées ce qui est est un défi scientifique connu.
Profil recherché
Nous cherchons un candidat curieux, s'intéressant à l'imagerie biomédicale, sachant travailler en autonomie aussi bien qu'en équipe, ayant un esprit ouvert et critique. Le candidat doit disposer des solides bases en mathématiques appliquées et programmation C++ (éventuellement Python) et de bonnes connaissances en traitement d'images. Les connaissances en apprentissage automatique sont un atout.
Candidature
Nous invitons le candidat intéressé à nous envoyer sa lettre de motivation, avec un CV et un relevé de notes récent.
Gratification
Le stagiaire sera rémunéré au tarif prévu par la loi ; ~ 540 € / mois.
Bibliographie
[KECH-14] R. Kéchichian, S. Valette, M. Sdika et M. Desvignes, “Automatic 3D multiorgan segmentation via clustering and graph cut using spatial relations and hierarchically-registered atlases,” MICCAI Medical Computer Vision: Algorithms for Big Data, 2014, pp. 201-209.
[KECH-13] R. Kéchichian, S. Valette, M. Desvignes et R. Prost, “Shortest-Path Constraints for 3D Multiobject Semiautomatic Segmentation via Clustering and Graph Cut,” IEEE Trans. on Image Processing, vol. 22, no. 11, pp. 4224-4236, 2013.
[BOYK-01] Y. Boykov, O. Veksler, and R. Zabih. "Fast approximate energy minimization via graph cuts." IEEE Transactions on pattern analysis and machine intelligence 23.11 (2001): 1222-1239.
[JIME-16] Jimenez-del-Toro, O., Muller, H., Krenn, M., Gruenberg, K., Taha, A. A., Winterstein, M., ... and Kontokotsios, G. (2016), "Cloud-based Evaluation of Anatomical Structure Segmentation and Landmark Detection Algorithms: VISCERAL Anatomy Benchmarks," in IEEE Transactions on Medical Imaging , vol.PP, no.99, pp.1-1, 2016.
[BISH-06] C. Bishop, Pattern Recognition and Machine Learning, Springer, 2006.
[TONG-15] Tong, T., Wolz, R., Wang, Z., Gao, Q., Misawa, K., Fujiwara, M., ... and Rueckert, D, "Discriminative dictionary learning for abdominal multi-organ segmentation," Medical image analysis, 23(1), 92-104, 2015.
[KOHL-11] Kohlberger, T., Sofka, M., Zhang, J., Birkbeck, N., Wetzl, J., Kaftan, J., ... and Zhou, S. K, "Automatic multi-organ segmentation using learning-based segmentation and level set optimization," In Intl. Conf. on Med. Image Computing and Computer-Assisted Intervention, pp. 338-345, 2011.
[SEIF-09] Seifert, S., Barbu, A., Zhou, S. K., Liu, D., Feulner, J., Huber, M., ... and Comaniciu, D. "Hierarchical parsing and semantic navigation of full body CT data," In SPIE medical imaging, pp. 725902-725902, 2009.
[ZHEN-15] L. Zheng, S. Duffner, K. Idrissi, C. Garcia and A. Baskurt. "Siamese Multi-layer Perceptrons for Dimensionality Reduction and Face Identification," Multimedia Tools and Applications, 2015.