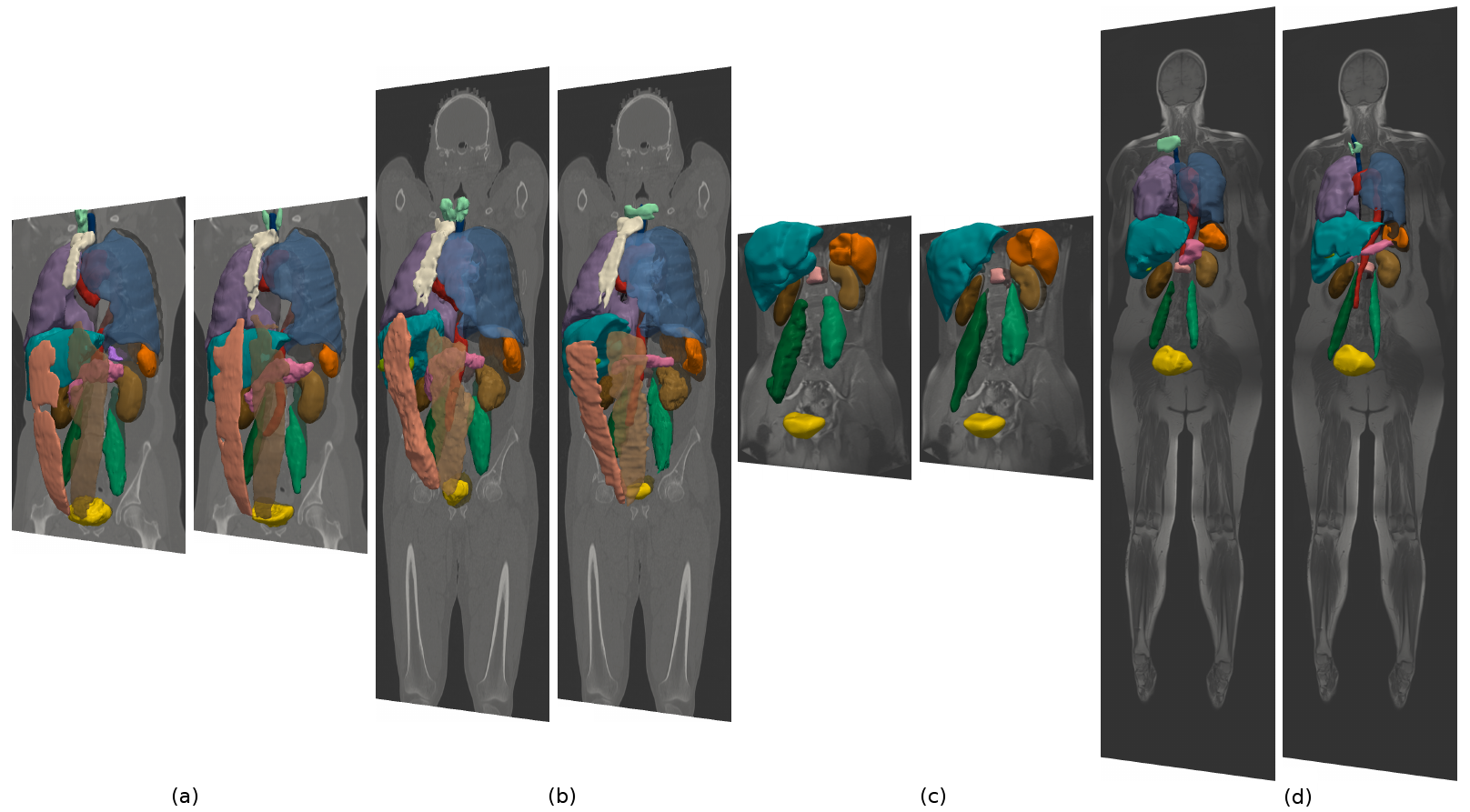
Illustration 1: Vues 3D des maillages surfaciques extraits des volumes de structures segmentées par notre méthode de segmentation multi-organe automatique. Dans chaque paire, l’image de gauche correspond à la segmentation, l’image de droite à la référence. (a) TDM thoracique-abdominale contrastée, (b) TDM non contrastée, (c) IRM abdominale contrastée et (d) IRM non contrastée.
Domaine et contexte scientifiques
Avec l’accumulation des grandes masses de données, en particulier des images médicales 3D de haute résolution de différentes modalités, les cliniques et les centres hospitaliers sont aujourd’hui confrontés au phénomène de “Big Data”. Les approches actuellement utilisées dans l’extraction des informations à partir des images tout au long de la chaîne clinique allant du diagnostic au suivi des patients reposent, pour la plupart, sur les outils d’analyse d’images classiques, souvent supervisés, qui ne se prêtent pas à un traitement automatique rapide et robuste de plusieurs milliers d’images. Ces limitations demandant des réflexions sur des nouvelles approches de traitement d’images, pouvant être sous-optimales, permettant d’extraire un maximum d’informations utiles en un temps réduit qui peuvent ensuite être complétées par des outils d’analyse et de traitement d’images plus poussés de type recalage et segmentation. Le vaste champ applicatif de ces méthodes couvre d’innombrables applications telles que l’aide au diagnostic, la radiothérapie, la création de modèles anatomiques patient-spécifiques, les études anthropométriques, la recherche d’images par le contenu, la création de prothèses sur-mesure à l’aide de l’impression 3D, l’éducation, etc.
Nous avons développé une méthode bayésienne de segmentation multi-organe automatique générique pour les images médicales 3D [KECH-14], formulée en termes d’une distribution a priori des relations d’adjacence entre les organes [KECH-13] et deux vraisemblances pour chaque organe : celle d’apparence, dérivée de l’image, et celle de position, représentée par une carte de probabilité spatiale construite, à partir d’une base d’apprentissage, sur l’image à l’aide de la méthode de recalage par points d’intérêt proposée par notre équipe [AGIE-16]. La segmentation optimale est obtenue en minimisant une énergie discrète à l’aide d’un algorithme d’optimisation par coupure de graphes multi-valuée [BOYK-1]. Notons que notre approche est classée parmi les 2 premières dans la segmentation de 20 organes thoraciques et abdominaux à partir des images TDM dans le “benchmark” des d’algorithmes de segmentation Visceral auquel nous participons depuis 2014. L’illustration 1 affiche quelques résultats obtenus à l’aide de cette méthode sur des images appartenant à 4 modalités différentes provenant de la base Visceral [LANG-12].
À partir de cette approche, ce projet se donne pour objectif d’aller au-delà et dès lors de proposer des avancées méthodologiques permettant d’aborder le problème du traitement et de l’extraction de connaissances à partir de grandes masses de données hétérogènes (Illustration 2), tout en autorisant un passage à l’échelle en termes de temps de calcul.

Illustration 2: 400 images TDM recalées dans un référentiel commun à l’aide de la méthode de recalage développée par notre équipe [AGIE-16].
Objectifs, verrous scientifiques, contributions originales attendues
Notre projet abordera, d’une part, le traitement automatique incrémental de grandes masses de données hétérogènes comportant des images entières ou partielles du corps à l’aide d’une approche rapide de détection du contenu, et, d’autre part, l’extraction plus robuste des connaissances à l’aide des méthodes d’apprentissage automa-tique et leur exploitation dans la segmentation de plusieurs dizaines d’organes par image dans un temps raisonnable.
Une détection robuste du contenu permet de vérifier la présence des structures dans une image 3D et de déterminer leurs positions approximatives rapidement. Ces informations, complétées par celles extraites de l’image peuvent ensuite être utilisées pour introduire des contraintes et des connaissances a priori dans l’algorithme de segmentation afin de délimiter les structures dans l’image précisément.
L’approche de segmentation développée s’appuiera sur la démarche décrite précédemment [KECH-14][KECH-13] et réalisera sa généralisation selon les axes suivants :
- Sélectionner et définir le modèle d’image (vraisemblances + a priori spatial) de manière automatique à l’aide de la détection du contenu : les méthodes de l’état de l’art en segmentation multi-organe [TONG-15, WOLZ-13, KOHL-11, SEIF-09, OKAD-15] s’appliquent à des images dont le contenu est connu au préalable.
- Mieux exploiter l’information d’image : Les vraisemblances d’apparence, se limitant dans notre approche aux simples histogrammes d’intensité, peuvent être mieux estimées à partir des descripteurs d’intensité et de texture à l’aide des méthodes d’apprentissage automatique. Cela nécessite une sélection et extraction automatiques des descripteurs adaptées à la modalité et à l’organe. Notons que les méthodes de l’état de l’art faisant intervenir l’apprentissage automatique [KOHL-11, SEIF-09] exploitent des descripteurs prédéfinis.
- Généraliser le modèle d’a priori de structure afin de tenir compte des relations d’orientation spatiale et des relations hiérarchiques : l’a priori de structure étendu servira à imposer des contraintes anatomiques plus puissantes sur l’espace de solutions conduisant à de meilleurs segmentations. Les méthodes de l’état de l’art adoptant une approche multi-atlas ou apprentissage automatique [WOLZ-13, OKAD-15] se limitent souvent à une simple régularisation spatiale binaire.
- Réduire le temps d’exécution de l’algorithme de segmentation afin de rendre son application à des cohortes d’images praticable : lié à des considérations comme le niveau de détail de la détection et la segmentation, il s’agit d’une question qui relève de l’optimisation des algorithmes et des représentations de données plutôt que de l’exploitation de davantage de ressources de calcul, bien que celles-ci soient indispensables.
Programme de recherche et démarche scientifique
Afin de répondre aux objectifs annoncés, les travaux de recherche se poursuivront selon deux axes interdépendants que nous détaillons ci-dessous.
Axe 1 - Détection du contenu d’images
Une approche de détection d’objets par points d’intérêt sera utilisée pour vérifier la présence des structures dans une image de manière automatique à l’aide des références dont les descripteurs seront extraits et stockés au préalable. Une même stratégie est également valable pour appliquer une méthode d’apprentissage automatique à l’aide des métriques ou des distributions apprises préalablement. Il serait alors intéressant de comparer les perfor-mances de ces deux approches. Il faudrait cependant s’adresser aux difficultés liées à chacune. Pour une approche par points d’intérêt, les liens entre les points détectés et les repères anatomiques doivent être établis, l’invariance à l’orientation 3D des descripteurs, ainsi qu’au contraste inter-modalités doivent être assurées pour une détection plus robuste sur des images de contenu et de modalité variables. Pour une approche d’apprentissage, les questions de choix de descripteur, de suffisance de données d’apprentissage et de généralisation des modèles appris doivent être pleinement adressées. Une méthode rapide de mise en correspondance par points d’intérêt SURF a récemment été développée par notre équipe [AGIE-16]. Elle a été appliquée avec succès au recalage déformable des cohortes de plusieurs centaines d’images TDM afin de les mettre dans un référentiel commun (Illustration 2) avec la détection des yeux comme une première application. Cette méthode constitue une piste très intéressante pour commencer les travaux de recherche sur la détection d’objets généralisée dans des cohortes d’images.
Axe 2 - Segmentation d’images par modélisation hiérarchique et apprentissage automatique
Sélectionner et définir le modèle d’image de manière automatique : notre approche est actuellement appli-quée aux images dont le contenu est connu au préalable. Dans une application sur des cohortes d’images où le con-tenu est inconnu, une approche de détection robuste du contenu permettra d’identifier les structures présentes dans l’image et de définir les vraisemblances et le modèle d’a priori spatial correspondant afin d’initialiser la segmentation.
Meilleure exploitation des informations de l’image : Les vraisemblances d’apparence évoquées plus haut seront estimées à partir des descripteurs d’intensité et de texture à l’aide des méthodes d’apprentissage automatique performantes comme les forêts aléatoires ou les réseaux de neurones avec une sélection et extraction automatique des descripteurs adaptées à la modalité de l’image et à l’organe. Les modèles de textures ou de formes locales peuvent être appris à l’aide des descripteurs classiques comme les histogrammes de gradients orientés (HOG) ou les “Local Binary Patterns” (LBP). Ces descripteurs peuvent également être appris à l’aide d’un réseau de neurones à convolution, soit de manière supervisée ou non-supervisée/supervisée mixte [ZHEN-15]. Les travaux visant à vérifier cet objectif sont déjà engagés par un stagiaire de recherche dans le cadre d’une collaboration avec l’équipe “Imagine” du laboratoire Liris (UMR 5205). Le projet poursuivra l’approfondissement et la généralisation des travaux entamés.
Extension du modèle d’a priori de structure : Outre les relations d’adjacence, un modèle de graphe pour les objets dans une image peut facilement représenter des relations d’orientation spatiale du type “le poumon droit se situe au-dessus du foie”. Ces relations nécessitent un graphe orienté, ce qui conduit à des contraintes asymétriques faisant appel à des modèles statistiques orientés [LAUR-01]. Par conséquent, l’algorithme d’optimisation par coupure de graphes multi-valuée doit être généralisé afin de tenir compte de ces contraintes. Un nouveau modèle d’image, représenté par des champs aléatoires conditionnels (CRF), constituera le cadre unificateur des axes de recherche détaillés jusqu’ici. Possédant l’avantage d’une puissance prédictive plus élevée, en particulier quand les modèles d’apparence sont apprises par des classifieurs performants et la capacité de modéliser les images selon différents niveaux conceptuels permettant des relations hiérarchiques [LUCC-11], les CRF présentent une alternative intéressante aux champs de Markov.
Amélioration des performances des algorithmes impliqués : en termes de durée d’exécution et d’empreinte mémoire afin de permettre le traitement de masses d’images en temps raisonnable. Plusieurs stratégies peuvent être adoptées : l’abandon des voxels au profit des partitions d’image ou, le cas extrême, une représentation par points d’intérêt très parcimonieuse ; une approche de détection et de segmentation incrémentale hiérarchique allant d’un faible niveau de détail anatomique vers un niveau plus élevé ; l’apprentissage incrémentale des distributions se rendant de plus en plus précises avec l’accumulation de données ; la parallélisation des algorithmes etc.
Données et évaluation : Nous conduisons nos recherches actuelles en segmentation sur la base de données multi-modalité Visceral [LANG-12] offrant 80 images de 4 modalités différentes où chaque image est accompagnée de 20 segmentations réalisées par des experts. Bien que de haute qualité, cette base est insuffisante pour bénéficier pleinement du potentiel des méthodes d’apprentissage automatique supervisées ainsi que pour conduire des évaluat-ions plus fiables des méthodes de détection et de segmentation. Dans l’objectif de constituer une base de données de taille suffisante, nous avons récemment établi un partenariat avec les centres hospitaliers universitaires de Nagoya et Osaka (Japon), qui disposent des bases de données comptant plusieurs centaines d’images médicales annotées. Ainsi, les travaux impliquant l’apprentissage automatique et la validation des méthodes seront menés avec la participation des partenaires internationaux. Le grand objectif de ces travaux de recherche est l’application des méthodes de détection et de segmentation sur une très grande base de données comptant des milliers d’images hétérogènes non annotées mise à notre disposition par les praticiens-hospitaliers membres de notre équipe de recherche.
Encadrement scientifique et collaboration internationale
Le candidat recruté intégrera l’équipe “Images et Modèles” du laboratoire Creatis et sera encadré par un comité composé de MM. Friboulet et Kéchichian. Responsable d’une équipe de recherche pluridisciplinaire, M. Friboulet dispose des connaissances en méthodologies appliquées à l’imagerie médicale et assurera le bon déroulement des travaux et leur valorisation. M. Kéchichian encadrera le candidat étroitement et apportera les connaissances liées aux méthodes de segmentation et d’optimisation, et, les travaux de recherche impliquant une importante dimension de données, les compétences spécifiques à l’implantation et la validation des méthodes proposées. Sur le plan méthodologique, la collaboration avec l’équipe “Imagine” du laboratoire Liris sur le thème d’extraction et d’exploitation de connaissances à partir des images sera maintenue et renforcée. Par ailleurs, comme évoqué plus haut, ce projet donne l’excellente opportunité d’entamer une collaboration internationale avec les centres hospitaliers universitaires de Nagoya et Osaka pour l’acquisition de nouvelles bases de données d’images médicales annotées. Au-delà du besoin de données de notre projet, cette collaboration contribuera au rayonnement internationale du laboratoire Creatis.
Profil du candidat recherché
Le candidat doit avoir un diplôme de M2 ou équivalent en sciences d’ingénieur. Il doit être curieux, s’intéresser à l’imagerie biomédicale, savoir travailler en autonomie aussi bien qu’en équipe, avoir un esprit ouvert et critique. Il doit en outre disposer des solides bases en mathématiques appliquées et programmation en C++ et Python, et de bonnes connaissances en traitement d’images. Les connaissances en apprentissage automatique sont un atout.
Objectifs de valorisation des travaux de recherche
Sur le plan de la valorisation scientifique, ces travaux de recherche seront soumis dans des revues internationales de premier rang (IEEE Trans. Image Proc., Int. J. Comput. Vision) et à plusieurs communications dans des conférences spécialisées de premier plan (IEEE ICIP, IEEE CVPR). À plus long terme, la valorisation de ces travaux se traduira sur le plan applicatif et sociétal par un déploiement de l’approche proposée en milieu clinique et son évalu-ation en termes de gain d’efficacité diagnostique. Une telle perspective est rendue possible par les liens privilégiés et les collaborations de longue dates établies entre Creatis et les Hospices Civils de Lyon, le CHU de Saint Étienne.
Compétences développées au cours de la thèse et perspectives professionnelles
Le candidat aura l’opportunité d’approfondir ses connaissances en traitement d’images et de développer de nouvelles compétences méthodologiques en apprentissage automatique et vision par ordinateur et des connaissances médicales en anatomie et imagerie. La pluridisciplinarité du projet contribuera non seulement à l’enrichissement de la culture scientifique du candidat mais aussi à l’élargissement de ses perspectives professionnelles dans le monde académique aussi bien qu’en industrie. À la suite du projet, le candidat pourra intégrer un laboratoire de recherche menant des travaux liés à une ou plusieurs thématiques abordées. Il pourra également intégrer un service R&D d’une entreprise de construction d’imageurs ou d’édition de logiciel et pratiquer un métier de “data scientist” ou de chargé de développement de logiciel de post-traitement.
Bibliographie
[KECH-14] R. Kéchichian, S. Valette, M. Sdika et M. Desvignes, “Automatic 3D multiorgan segmentation via clustering and graph cut using spatial relations and hierarchically-registered atlases,” MICCAI Medical Computer Vision: Algorithms for Big Data, 2014, pp. 201-209.
[KECH-13] R. Kéchichian, S. Valette, M. Desvignes et R. Prost, “Shortest-Path Constraints for 3D Multiobject Semiautomatic Segmentation via Clustering and Graph Cut,” IEEE Trans. on Image Processing, vol. 22, no. 11, pp. 4224-4236, 2013.
[LANG-12] Langs, G., Hanbury, A., Menze, B., & Müller, H. (2012, October). Visceral: Towards large data in medical imaging—challenges and directions. In MICCAI International Workshop on Medical Content-Based Retrieval for Clinical Decision Support (pp. 92-98). Springer Berlin Heidelberg.
[AGIE-16] R. Agier, S. Valette, L. Fanton, P. Croisille, and R. Prost, "Hubless 3D medical image bundle registration", VISAPP ’16.
[BOYK-01] Y. Boykov, O. Veksler, and R. Zabih, “Fast approximate energy minimization via graph cuts,” IEEE Trans. Pattern Anal. Mach. Intell., vol. 23, no. 11, pp. 1222–1239, 2001.
[LAUR-01] S. L. Lauritzen, “Causal inference from graphical models. Monographs on Statistics and Applied Probability,” vol. 87, pp. 63-108, 2001.
[LUCC-11] A. Lucchi, Y. Li, X. Boix, K. Smith, and P. Fua, “Are spatial and global constraints really necessary for segmentation?” in IEEE Int. Conf. on Computer Vision, Nov. 2011, pp. 9–16.
[TONG-15] Tong, T., Wolz, R., Wang, Z., Gao, Q., Misawa, K., Fujiwara, M., ... & Rueckert, D. (2015). Discriminative dictionary learning for abdominal multi-organ segmentation. Medical image analysis, 23(1), 92-104.
[WOLZ-13] Wolz, R., Chu, C., Misawa, K., Fujiwara, M., Mori, K., & Rueckert, D. (2013). Automated abdominal multi-organ segmentation with subject-specific atlas generation. IEEE Trans on Med. imaging, 32(9), 1723-1730.
[KOHL-11] Kohlberger, T., Sofka, M., Zhang, J., Birkbeck, N., Wetzl, J., Kaftan, J., ... & Zhou, S. K. (2011, September). Automatic multi-organ segmentation using learning-based segmentation and level set optimization. In Intl. Conf. on Med. Image Computing and Computer-Assisted Intervention (pp. 338-345), Springer.
[SEIF-09] Seifert, S., Barbu, A., Zhou, S. K., Liu, D., Feulner, J., Huber, M., ... & Comaniciu, D. (2009, February). Hierarchical parsing and semantic navigation of full body CT data. In SPIE medical imaging (pp. 725902-725902).
[OKAD-15] Okada, T., Linguraru, M. G., Hori, M., Summers, R. M., Tomiyama, N., & Sato, Y. (2015). Abdominal multi-organ segmentation from CT images using conditional shape–location and unsupervised intensity priors. MedIA, 26(1), 1-18.
[ZHEN-15] Lilei Zheng, Stefan Duffner, Khalid Idrissi, Christophe Garcia & Atilla Baskurt (2015). “Siamese Multi-layer Perceptrons for Dimensionality Reduction and Face Identification”. Multimedia Tools and Applications.
