Contexte :
Le sujet de stage s’inscrit dans le projet de recherche IMAGINA financé par l'ANR et regroupant des spécialistes aux expertises complémentaires en analyse d’images médicales, en particulier par apprentissage (machine learning)(CREATIS), en neurosciences computationnelles (CRNL), en médecine (HCL) et en acquisition d’images et traitement (CERMEP). L'un des objectifs de ce projet est de développer des outils de prédiction automatisés par apprentissage qui évaluent l'état de coma des patients atteints de troubles de la conscience en combinant les informations fournies par l'imagerie multimodale in vivo (IRM, TEP).
L’originalité de ce projet est de réaliser les acquisitions sur une machine hybride IRM-TEP permettant l'acquisition simultanée de l'information fonctionnelle par tomographie par émission de positons (TEP), et des informations fonctionnelles ou structurelles par imagerie par résonance magnétique (IRM). Les deux modalités sont complémentaires : la TEP offre une sensibilité inégalée à un niveau moléculaire, alors que l'IRM offre un excellent contraste des tissus mous ainsi qu’un certain nombre d’informations supplémentaires (diffusion de l’eau, circulation sanguine, tractographie, spectroscopie). L’acquisition simultanée est un moyen unique pour comprendre les interdépendances et les interactions des fonctions physiologiques.
Sujet du stage :
L’objectif du stage est de développer un modèle prédictif de l’état de conscience par apprentissage permettant l’analyse des données IRM-TEP de la cohorte unique de sujets comateux en phase aigüe (env. 100 examens) acquise dans le cadre du projet IMAGINA. La taille de la cohorte étant limitée (100 patients) par rapport au volume de données collectées pour chaque patient (>10 volumes d’images 3D), nous privilégierons, pour cette première étude, une approche par apprentissage classique, consistant d’une part à extraire l’information pertinente de chaque examen puis à entrainer un modèle statistique sur les caractéristiques extraites.
Déroulement du stage :
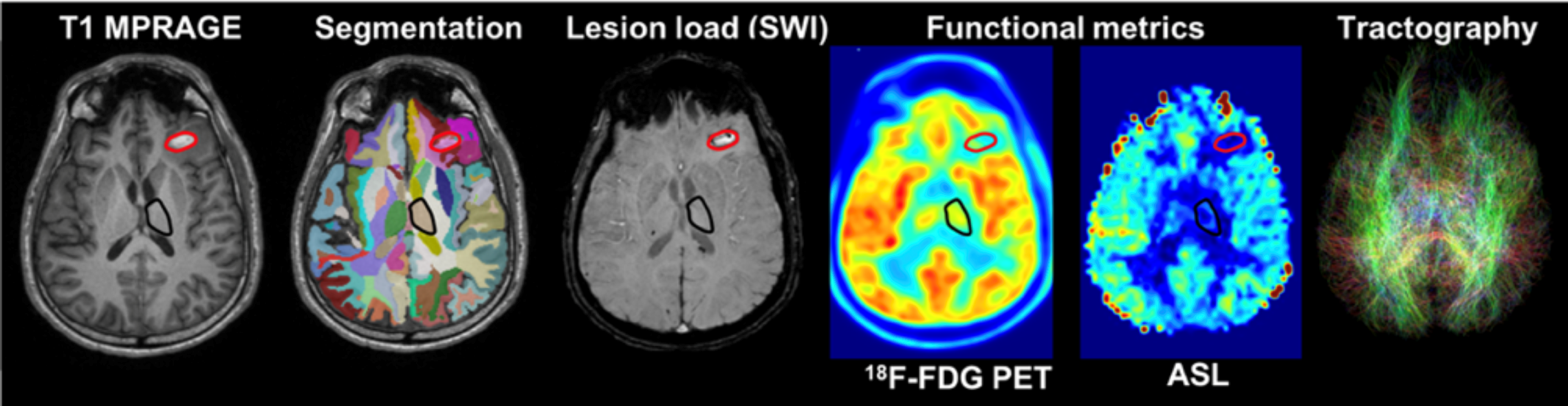
Les images brutes acquises sur la machine IRM-TEP doivent être soumises à une chaine de traitement afin de transformer la nature physique du signal en paramètre biologique, sous forme d’image paramétrique. Les méthodes de pré-traitement, traitement des images TEP et IRM sont déjà choisies et intégrées dans des scripts.
Les méthodes d’intelligence artificielle seront choisies parmi les méthodes classiques d’apprentissage par machine. Les différentes tâches à réaliser au cours du stage sont les suivantes :
- Prise de connaissance des objectifs de l’étude et des méthodes d’analyse, lecture des articles scientifiques de référence
- Prise en main et exécution des scripts de traitement sur des données réelles acquises sur l’imageur IRM-TEP dans le cadre de l’étude IMAGINA
- Pré-traitements des différentes modalités IRM (IRM de diffusion, IRM de perfusion ASL) et réalisation de leur cartes paramétriques (FA, MD, perfusion)
- Segmentation régionale des images anatomiques IRM (T1, FLAIR)
- Modélisation des images dynamiques TEP au [18F]FDG pour la réalisation des cartes paramétriques de consommation de glucose cérébrale (CMRGlu)
- Normalisation spatiale des images anatomiques (T1, FLAIR, T2*, SWI…) et des cartes paramétriques (FA, MD, perfusion, CMRGlu) dans un espace standardisé
- Extraction des valeurs régionales des cartes paramétriques sur une sélection de régions d’intérêt
- Pré-traitements des différentes modalités IRM (IRM de diffusion, IRM de perfusion ASL) et réalisation de leur cartes paramétriques (FA, MD, perfusion)
- Mise en forme des vecteurs de caractéristiques multiparamétriques régionaux pour l’apprentissage et analyse des distributions de ces caractéristiques pour les différentes classes de patients.
- Sélection et implémentation de différents algorithmes d’apprentissage statistique (e.g. SVM, réseau de neurones, arbre de décision..)
- Evaluation des performances de classification : choix des métriques de performance et de la stratégie d’évaluation (e.g. validation croisée).
- Analyses statistiques des résultats
- Implémentation d’une méthode post hoc d’explicabilité permettant d’extraire les caractéristiques les plus influentes de la tâche de classification.
- Implémentation de méthodes de mesure d’incertitude permettant de quantifier le niveau de confiance du modèle prédictif.
Les deux dernières tâches listées ci-dessus sont optionnelles. Leur mise en œuvre sera discutée en fonction des compétences du ou de la candidat.e et de la durée du stage.
Le candidat / la candidate :
Ce stage convient à un.e étudiant.e en ingénierie informatique, génie électrique ou génie biomédical possédant des bases solides en programmation et des connaissance en traitement et analyse d’images et en apprentissage statistique.. Nous recherchons un ou une étudiant.e enthousiaste et autonome, fortement motivé.e et intéressé.e par la recherche multidisciplinaire (traitement d'images et apprentissage automatique dans un contexte médical).
L’encadrement :
L'étudiant sera encadré par Nicolas Costes (PhD) et Inés Mérida (PhD), ingénieur.e.s de recherche sur la plateforme IRM-TEP au CERMEP et Carole Lartizien (PhD), directrice de recherche à CREATIS.
Le stage se déroulera au CERMEP - Imagerie du vivant (Centre d’Etude Multimodal et Pluridisciplinaire, http://www.cermep.fr/), Pôle Hospitalier Lyon Est, 59 bd Pinel, Lyon 3ème.
Le sujet de ce stage est prévu pour un travail d’une durée de 4 à 6 mois
Informations et contact :
Pour candidater envoyer un CV ainsi qu’une lettre de motivation par voie électronique.
