Accueil : Laboratoire CREATIS (INSA Lyon, 69100 Villeurbanne) – Equipe ULTIM/ MYRIAD
Encadrants : Philippe Delachartre (philippe.delachartre@creatis.insa‐lyon.fr )
Valentine Warnier Dauchelle (valentine.wargnier@creatis.insa-lyon.fr )
Carole Lartizien (carole.lartizien@creatis.insa-lyon.fr)
Keywords : Deep Learning, Computer Vision, Detection, Segmentation, Medical Imaging, Supervised and semi-supervised Approaches
Durée : 5-6 mois.
Période de démarrage : Février-mars 2026
Rémunération : ~ 600 euros/mois
Contexte
Nous avons développé une nouvelle méthode d’imagerie échographie 3D pour l’enfant prématuré [1] permettant notamment de visualiser le développement des structures cérébrales au cours du temps. Par rapport à l’IRM, l’examen échographique présente l’avantage d’être réalisé au lit de l’enfant, il est également applicable à tous les enfants car chaque centre de néonatologie possède un échographe et l’examen peut même être répété 3 à 5 fois pendant le séjour de l’enfant. C’est ce dernier point que nous voulons développer pour suivre l’évolution des lésions cérébrales de la matière blanche (PWML) car aucun outil n’existe à ce jour en échographie. Dans ce projet, nous nous intéresserons principalement à l’évolution des lésions et des thalami de la matière blanche sur des enfants ayant eu 3 examens échographiques définis aux 3 temps SA33, SA35 et SA37 (SA : Semaines d’Aménorrhées).
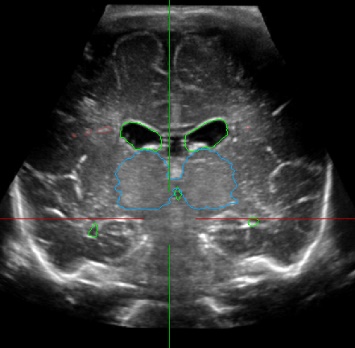
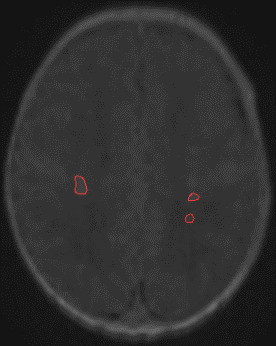
Figure 1: (a) and (b) Manual segmentation of thalami (blue), CVS (green) and PWML (red) in a reconstructed 3D cUS acquisition of a premature infant, (c) 3D cUS manual segmentation and (d) corresponding 3D MRI automatic segmentation of PWMLs (red) in the same premature infant.
Objectif
Le but de ce stage est d’étudier puis de développer des modèles d’apprentissage automatique pour la détection-segmentation des lésions cérébrales (PWML figure 1) et leur suivi sur plusieurs examens, en utilisant des modèles supervisés pour la segmentation (ex : TransU-Net [2], Swin-UNet [3] ou Mask R-CNN [4]) et entrainés pour chaque temps d’examen (approche statique supervisée). Dans l’idée d’alléger l’annotation du médecin pour chaque structure, on propose d’aborder une approche dynamique semi-supervisée inspirée des méthodes de segmentation de séquences vidéos [5]. Ces méthodes dites VOS (Video Object Segmentation) basées sur les transformeurs ont été développées pour la mise en correspondance des pixels entre l’image (frame) de référence avec l’image cible (Query frame) atteignant de très bonnes performances [5].
Tout au long du processus de développement, une démarche d’IA écoresponsable sera suivie [6].
Déroulement
1/ Bibliographie, compréhension des méthodes actuelles d’apprentissage supervisées et semi-supervisées pour la segmentation de structures cérébrales. Compréhension et analyse des images de la base de données.
2/ Développement et programmation de 2 méthodes adaptées à l’application visée. On commencera par appliquer les méthodes choisies sur les images, puis sur des volumes en fonction de l’avancement.
3/ Validation sur la base de données d’échographie 3D de CREATIS.
4/ Rédaction d’un document de synthèse en anglais (rapport) et rédaction d’une publication en fonction de l’avancement.
Compétences requises
Machine et Deep Learning, détection d’anomalies, segmentation
Langages de programmation et librairies : Python, Tensorflow/PyTorch, Scikit-Learn
Environnement de travail : Linux, Windows
Autonomie et goût pour le travail dans un environnement interdisciplinaire
Encadrement et contacts
Philippe Delachartre (delachartre@creatis.insa-lyon.fr )
Valentine Warnier Dauchelle (valentine.wargnier@creatis.insa-lyon.fr )
Carole Lartizien (carole.lartizien@creatis.insa-lyon.fr)
[1] Matthieu Martin, Bruno Sciolla, Michaël Sdika, Philippe Quétin, Philippe Delachartre, Automatic segmentation and location learning of neonatal cerebral ventricles in 3D ultrasound data combining CNN and CPPN, Computers in Biology and Medicine, Volume 131, 2021, 104268, ISSN 0010-4825, https://doi.org/10.1016/j.compbiomed.2021.104268.
[2] F. Estermann, V. Kaftandjian, P. Guy, P. Quetin and P. Delachartre, " CACTUS: Multiview Classifier for Punctate White Matter Lesions Detection & Segmentation in Cranial Ultrasound Volumes," Computers in Biology and Medicine, Volume 197, Part B, 2025, 111085, ISSN 0010-4825, https://doi.org/10.1016/j.compbiomed.2025.111085.
[3] Cao, Hu, et al. "Swin-unet: Unet-like pure transformer for medical image segmentation." European conference on computer vision. Cham: Springer Nature Switzerland, 2022.
[4] He, Kaiming, et al. "Mask r-cnn." Proceedings of the IEEE international conference on computer vision. 2017.
[5] Ho Kei Cheng, Seoung Wug Oh, Brian Price, Joon-Young Lee, Alexander Schwing; Putting the Object Back into Video Object Segmentation. Proceedings of the IEEE/CVF Conference on Computer Vision and Pattern Recognition (CVPR), 2024, pp. 3151-3161.
[6] Dilhac, M.‐A., Abrassart, C., Voarino, N., et al. (2018b). Rapport de la déclaration de Montréal pour un développement responsable de l’intelligence artificielle. Technical report, IA responsable.
Présentation du laboratoire CREATIS :
Le laboratoire Creatis est une Unité de recherche en imagerie médicale regroupant environ 200 personnes (58 chercheurs et enseignants-chercheurs, 11 chercheurs et 18 médecins des Hospices Civils de Lyon (HCL) et du CHU de Saint Etienne, 75 doctorants et Post-doctorants, 22 ingénieurs, des informaticiens, techniciens et personnels administratifs) dont les domaines de recherche privilégiés sont au croisement de deux grands axes :
• L’identification des grandes questions de Santé pouvant être abordées par l'Imagerie.
• L’identification des verrous théoriques en traitement du signal & des images, en modélisation & en simulation numérique dédiés à l’imagerie du vivant.
Creatis répond à ces défis par une approche transdisciplinaire reposant sur une organisation matricielle mettant en interaction ses six équipes de recherche appartenant aux sciences et technologies de l’information et de la communication, aux sciences pour l’ingénieur et aux sciences du vivant.
