- Participants
P. Clarysse, N. Duchateau (eq. 1), G. K. Rumindo (PhD student)
P. Croisille, M. Viallon (eq. 5)
- Collaborations nationales et internationales
J. Ohayon (TIMC-IMAG, Grenoble, France)
P. Bovendeerd (TU/e, Eindhoven, Pays Bas)
V. Wang, M. Nash, A. Young (Auckland Bioengineering Institute, Nouvelle Zélande)
- Question
Comment caractériser avec précision, suivre et prédire l’évolution de la récupération fonctionnelle cardiaque suite à une revascularisation coronarienne ?
- Objectif
Notre approche est basée sur la personnalisation de modèles par éléments finis 3D (EF-3D) de cœur battant à partir de données issues d’une imagerie dynamique. L’état fonctionnel du cœur est étudié au travers des paramètres mécaniques des modèles. Ces derniers sont analysés statistiquement sur des populations de modèles EF-3D, identifiés à partir de données cliniques ou créés artificiellement, pour déterminer ceux qui caractérisent le mieux la pathologie. Il s’agit donc de:
- Personnaliser un modèle EF-3D de cœur battant à partir de données issues d’une imagerie cardiaque dynamique,
- Générer des populations de modèles avec des caractéristiques différentes en termes de propriétés mécaniques et de pathologie,
- Identifier les paramètres caractérisant la fonction myocardique et son évolution au décours de la maladie et suite à la revascularisation.
- Méthode
- Personnalisation par une approche bio-mécanique inverse. Le modèle EF-3D est basé sur la loi de comportement transverse isotrope et quasi incompressible de Fung adaptée au myocarde par Guccione et al. pour la partie passive et une loi de force active dépendante de l’étirement. L’implantation EF est réalisée sous Abaqus. La personnalisation résulte d’une optimisation non linéaire sous contrainte de la loi pression-volume et/ou de déformations mesurées en imagerie,
- Génération de population de modèles. A partir d’une base de modèles géométriques, de nouveaux cas sont créés par tirage statistique dans la gamme de variation des paramètres mécaniques et d’infarctus,
- Identification des marqueurs de la fonction. Une analyse de données statistique et intégrative est conduite sur des cohortes de cas de synthèse et cliniques pour la détermination des paramètres caractérisant les propriétés mécaniques passives et actives du myocarde (cf. action ‘analyse de paramètres et extraction de marqueurs.
- Résultats et illustrations
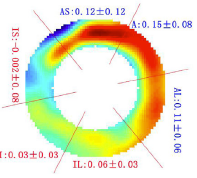
La Figure 1 illustre quelques exemples de modèles EF-3D de ventricule gauche (VG) avec des zones lésionnelles de forme, de taille, de localisation et de paramètres mécaniques différents issus de notre base de modèles de synthèse [RUMI-17], [RUMI-17a].
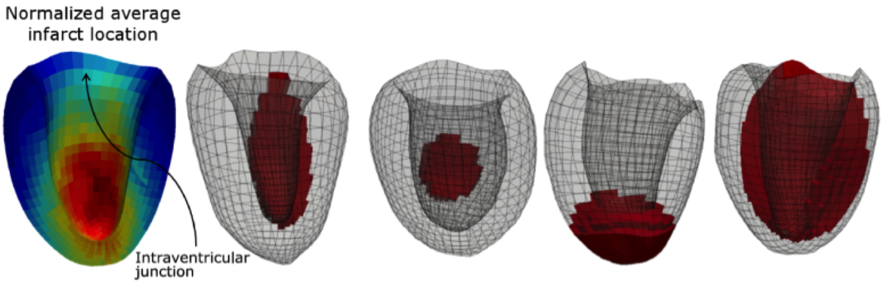
Figure 1. Distribution moyenne des lésions dans une base de 200 cas de VG pathologiques de synthèse (gauche, la zone pathologique la plus représentée étant en rouge) et quatre exemples de lésions de différente forme, taille et emplacement.
La Figure 2 montre les cartographies de 8 paramètres classiques de déformation produites à partir d’un modèle EF-3D pour lequel la pathologie a la configuration géométrique et mécanique indiquée. On peut voir notamment que les déformations principale, radiaire et certains invariants localisent clairement la lésion [RUMI-17a]. La déformation selon les fibres myocardiques (ang. fiber strain) a été plus particulièrement étudiée en croisant des données fonctionnelles (IRM marquée) et des données architecturales (IRM de tenseur de diffusion) dans le cadre d’une collaboration avec le groupe de recherche cardiaque à l’Auckland Bioengineering Institute [WANG-16].
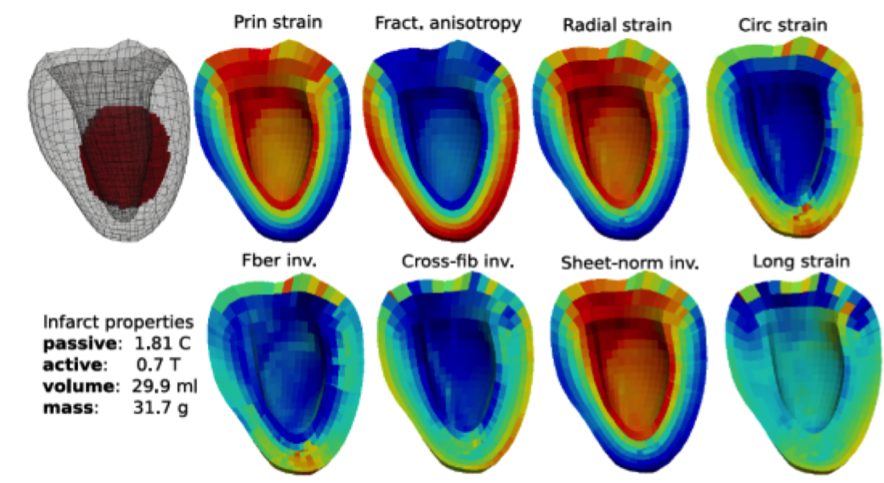
Figure 2. Cartographie 3D de 8 paramètres de déformation issue d’un modèle EF-3D de VG avec zone lésionnelle où la raideur est 1.8 fois supérieure à la valeur normale et l’activation réduite à 70%.
- Soutiens financiers
- Projet Marie-Curie VPH-CaSE (VPH-Cardiovascular Simulation and Experimentation for Personalised Medical Devices, H2020-MSCA-ITN-2014) : financement thèse G. K. Rumindo.
- Projet PALSE-IMPULSION CardioMIX (Mixing and learning multiple heterogeneous descriptors of cardiac function : integrating clinical constraints and expectations), N. Duchateau.
- Projet ANR MOSIFAH (Modélisation et simulation multimodales et multiéchelles de l'architecture des fibres myocardiques du cœur humain, ANR-13-MONU-0009), Y. Zhu.
- Références bibliographiques
- Journaux
[WANG-16] V. Wang, C. Casta, Y. M. Zhu, B. Cowan, P. Croisille, A. Young, P. Clarysse, and M. Nash, "Image-based investigation of human in vivo myofibre strain," IEEE Transactions on Medical Imaging, vol. PP, no. 99, pp. 1-1, 2016.
- Conférences
[RUMI-16] G. K. Rumindo, J. Ohayon, M. Viallon, M. Stuber, P. Croisille, and P. Clarysse, "Comparison of different strain-based parameters to identify human left ventricular myocardial infarct: a three-dimensional finite element study," Computer Methods in Biomechanics and Biomedical Engineering (CMBBE), Tel-Aviv, Israel, 2016.
[RUMI-17] G. K. Rumindo, N. Duchateau, P. Croisille, J. Ohayon, and P. Clarysse, "Strain-Based Parameters for Infarct Localization: Evaluation via a Learning Algorithm on a Synthetic Database of Pathological Hearts," in Functional Imaging and Modeling of the Heart (FIMH), Toronto, Canada, 2017, pp. 106-114.
[RUMI-17a] G. K. Rumindo, N. Duchateau, M. Viallon, P. Croisille, J. Ohayon, and P. Clarysse, "Generation of a synthetic database of heart models to evaluate stran-based infarct detection algorithms," in Recherche en Imagerie et Technologies pour la Santé, Lyon, France, 2017.